SBMLによる生化学ネットワークの記述と計算
SBMLは、Systems Biology Markup Languageの略。シグナル伝達や代謝系などの生化学ネットワークのモデルを形式的に記述するXML仕様で、2000年頃から継続的に策定と改訂が行われているらしい。生物分野で古典的な数理モデルといえば、酵素反応速度論(Michaelis-Menten式etc.)、Hodgkin-Huxley方程式、Turingの反応拡散系などがあるけど、前の2つは、SBMLで記述されるモデルのプロトタイプと言っていいと思う。これらのモデルは、数個の変数しかない単純なモデルだけど、最近では細胞内の生化学ネットワークを調整する遺伝子たちが沢山同定されて、複雑な生化学ネットワークを、そのままモデル化して調べるという道ができた。
その極致として、2012年に、500個の遺伝子を持つ細菌マイコプラズマの全細胞シミュレーションが行われている
A whole-cell computational model predicts phenotype from genotype.
https://www.ncbi.nlm.nih.gov/pubmed/22817898
いずれ、線虫(細胞数1000、遺伝子数20000弱)くらいの生物で全細胞シミュレーションができれば面白いと思うけど、全ての要素をモデル化するだけでも大変そう(マイコプラズマの場合でも、シミュレーションの結果から新しい酵素が見つかったとかあったようだし)
#関連する話題として、最近、RIKENで同じくマイコプラズマの全原子分子動力学シミュレーションを行ったという報告があった
バクテリア細胞質の全原子分子動力学計算(プレスリリース)
http://www.riken.jp/pr/press/2016/20161101_3/
こっちは、20nsを数カ月かけて計算するというものらしいので、計算量的に何かへ応用するというには、難しいけれど。
More Detailed Summary of SBML
http://sbml.org/More_Detailed_Summary_of_SBML
に、もう少し詳しいSBMLの説明と、ミカエリス・メンテン型酵素反応の例が載っている。
既に知られているモデルの場合は、SBMLを
BioModels Database
http://www.ebi.ac.uk/biomodels-main/
とかから見つけてこれるかもしれない。Curatedでないモデルは色々ダメなことが多いっぽいので、使えるモデルは600ちょっとと思っておいたほうがよさげではある。まぁ、この手のシミュレータを実装すること自体は、難しいことでもないと思うけど、何気にパラメータが多くて、論文などから適切な値を探すのが面倒だったりするので、モデルをまとめて公開してくれているとありがたい。
SBMLのシミュレータはいくつも公開されている。
SBML Software Matrix
http://sbml.org/SBML_Software_Guide/SBML_Software_Matrix
にソフトウェアのリストがある。libSBMLとかあるけど、こいつにはシミューレション機能とかはなく、SBMLファイルの読み書きをするだけで、割とどうでもいい。対応しているSBMLのバージョン、実装されている機能や計算速度、継続的にメンテナンスがされているのかなどを考慮して決めたいのだけど、いまいちよく分からないので、自分で実装した方が早そうであった
最も単純な利用法は、常微分方程式系に変換してソルバーに投げるというのだと思う。細胞は非常に小さく、分子種によっては分子数が非常に少ない場合があり、ゆらぎが無視できない場合もあると考えられており、その場合は確率モデルを用いる必要があると考えられている。その他にも、目的に応じて、適用したい解析手法は多数あると思う。
自分でシミュレータを実装する場合は
SBML Test Suite
http://sbml.org/Software/SBML_Test_Suite
から、テストデータやらを引っ張ってこれるっぽいので、確認用に使える。
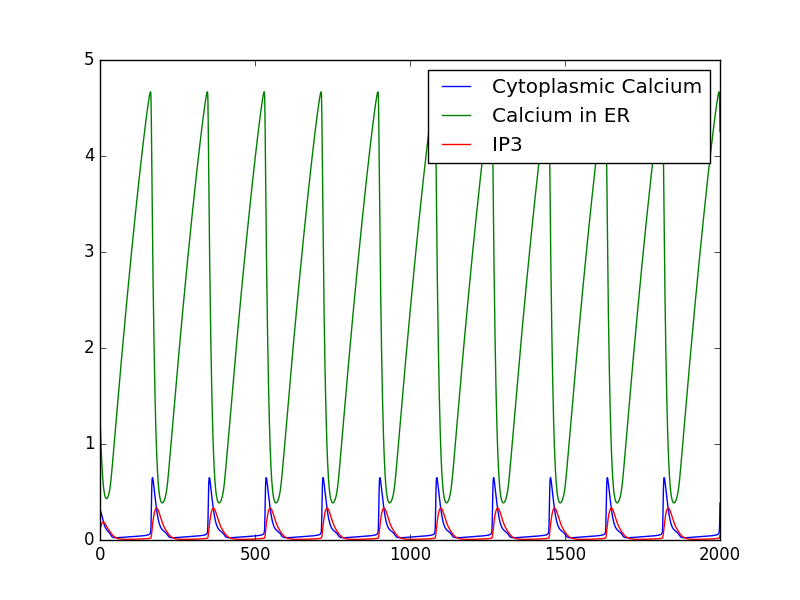
例として、カルシウム振動のモデル
http://www.ebi.ac.uk/compneur-srv/biomodels-main/BIOMD0000000184
を使う。アストロサイトにおけるカルシウム振動の(多分最も単純な)モデル。変数としては細胞質のカルシウム濃度とIP3(イノシトール3リン酸)濃度と、ERのカルシウムの濃度。
(1)細胞外から細胞内へのCa2+の流入及び流出
(2)ERから細胞質へのカルシウムの放出(IP3受容体を介するもの)
(3)細胞質からERへのカルシウムの浸透
(4)ERから細胞質へのカルシウムの漏れ
(5)IP3の合成(カルシウム濃度依存。ホスホリパーゼCの活性がカルシウムによる調節を受けるらしい)
(6)IP3の分解
が考慮される。3つの変数が振動を起こすことが確認できる。計算結果では、振動周期は3分程度である。これが、カルシウム振動のモデルとして適切なのかどうかは不明
とりあえず、これくらいのモデルがPython(+numpy/scipy/matplotlib)で動くようにした
・多分、2.7と3.5の両方で動く
・Test Suiteは通してないし、仕様の確認もしてないので、仕様を網羅もしてないと思う
・凄く遅い
いうて、常微分方程式やし、簡単にできると思ったが、色々面倒くさいことがわかった。とりあえずミカエリス・メンテン型反応とカルシウム振動の計算結果を貼っておく
from scipy.integrate import ode
import xml.etree.ElementTree as et
import math
from numpy import prod
def sbml_root_fn(xs):
if len(xs)==1:
return math.sqrt(xs[0])
elif len(xs)==2:
return math.pow(xs[1] , 1.0/xs[0])
else:
assert(False),"invalid argument in sbml_root_fn:{}".format(str(xs))
class Closure:
def __init__(self , _vars , fnbody):
self.vars = _vars
self.fndef = fnbody
self.env = {}
def __call__(self , args):
assert(len(args)==len(self.vars)),"invalid closure call"
saved_env = {}
for (key,val) in zip(self.vars , args):
if key in self.env:
saved_env[key] = self.env[key]
self.env[key] = val
ret = eval_math(self.fndef , self.env)
for key in self.vars:
del self.env[key]
for (key,val) in saved_env.items():
self.env[key] = val
return ret
def parse_math(e):
def parse_cn(e):
assert(e.attrib.get('type',None)!="rational"),"rational cn@parse_cn"
return eval(e.text)
def parse_apply(es):
fn = None
args = []
for e0 in es:
_ , tagname = e0.tag.split('}')
if tagname=="plus":
fn = (lambda x:x[0]+x[1])
elif tagname=="minus":
fn = (lambda x:x[0]-x[1])
elif tagname=="divide":
fn = (lambda x:x[0]/x[1])
elif tagname=="times":
fn = (lambda x:prod(x))
elif tagname=="power":
fn = (lambda x:math.pow(x[0],x[1]))
elif tagname=="exp":
fn = (lambda x:math.exp(x[0]))
elif tagname=="root":
fn = sbml_root_fn
elif tagname=="apply":
args.append( parse_math(e0) )
elif tagname=="degree":
assert(fn==sbml_root_fn)
assert(len(e0.getchildren())==1),"error in parse_apply@degree"
e1 = e0.getchildren()[0]
_ , tagname1 = e1.tag.split('}')
if tagname1=="ci":
args.append( e1.text.strip() )
elif tagname1=="cn":
assert(e0.attrib.get('type',None)!="rational"),"rational cn@parse_math"
args.append( eval(e1.text) )
else:
assert(False),"failed to parse degree"
elif tagname=="ci":
if fn!=None:
args.append( e0.text.strip() )
else:
fn = e0.text.strip()
elif tagname=="cn":
assert(e0.attrib.get('type',None)!="rational"),"rational cn@parse_math"
args.append( eval(e0.text) )
else:
assert(False),"@parse_math:unknown tag {}".format(tagname)
assert(fn!=None),et.tostring(es)
return (fn,args)
def parse_lambda(es):
varnames = []
for e0 in es:
_ , tagname = e0.tag.split('}')
if tagname=="bvar":
for it in e0.getchildren():
varnames.append( it.text.strip() )
elif tagname=="apply":
fnbody = parse_apply(e0.getchildren())
return Closure(varnames , fnbody)
else:
assert(False),"unknown tag name@parse_lambda:{}".format(tagname)
_ , tagname = e.tag.split('}')
if tagname=="math":
assert(len(e.getchildren())==1),"error in parse_math"
return parse_math(e.getchildren()[0])
elif tagname=="lambda":
return parse_lambda(e.getchildren())
elif tagname=="apply":
return parse_apply(e.getchildren())
elif tagname=="cn":
return parse_cn(e)
else:
assert(False),"unknown tagname@parse_math:{}".format(tagname)
def eval_math(e , env):
if type(e)==tuple:
fn,_args = e
args = [eval_math(x,env) for x in _args]
if type(fn)!=str:
return fn(args)
else:
closure = env[fn]
closure.env = env
return closure(args)
elif type(e)==str:
if e in env:
return env[e]
else:
assert(False),"Unknown variable in env:{}".format(e)
else:
return e
class Model:
def __init__(self):
self.args = []
self.species = []
self.namemap = {}
self.env = {}
self.reactions = []
self.compartment = {}
self.rules = {}
def values(self):
ret = []
for key in self.args:
v = self.env[key]
if type(v)==float or type(v)==int:
ret.append( self.env[key] )
else:
ret.append( eval_math(v ,self.env) )
return ret
def __call__(self , t , y):
assert(len(y)==len(self.args)),"invalid input"
ret = [0.0 for _ in y]
for (name,val) in zip(self.args , y):
self.env[name] = val
rest = self.rules.keys()
Nmax = len(rest)
#-- process assignment rules
for _ in range(Nmax):
if len(rest)==0:break
for (key,sm) in self.rules.items():
if key in rest:
try:
self.env[key] = eval_math(sm , self.env)
rest.remove(key)
except:
pass
#-- process reactions and rate rules
for (reactants , products , sm , local_env) in self.reactions:
saved_env = {}
for key,val in local_env.items():
if key in self.env:
saved_env[key] = self.env[key]
if type(val)==str:
self.env[key] = self.env[val]
else:
self.env[key] = val
try:
v = eval_math(sm , self.env)
except:
assert(False),("failed to evaluate math expresion",sm)
for r in reactants:
try:
idx = self.args.index(r)
if r in self.compartment:
ret[idx] -= v/self.env[self.compartment[r]]
else:
ret[idx] -= v
except:
pass
for r in products:
try:
idx = self.args.index(r)
if r in self.compartment:
ret[idx] += v/self.env[self.compartment[r]]
else:
ret[idx] += v
except:
pass
for key in local_env:
del self.env[key]
for key,val in saved_env.items():
self.env[key] = val
return ret
def readsbml(root):
ns = {'sbml' : root.tag[1:-5] , 'mml':"http://www.w3.org/1998/Math/MathML"}
ret = Model()
for it in root.find('.//sbml:listOfCompartments' , namespaces=ns).getchildren():
key,value = it.attrib['id'] ,float( it.attrib['size'] )
ret.env[key] = value
for it in root.find('.//sbml:listOfSpecies' , namespaces=ns).getchildren():
if it.attrib.get('boundaryCondition' , 'false')!='true':
ret.args.append( it.attrib['id'] )
if 'name' in it.attrib:
ret.species.append( it.attrib['name'] )
ret.namemap[ it.attrib['id'] ] = it.attrib['name']
else:
ret.species.append( it.attrib['id'] )
ret.namemap[ it.attrib['id'] ] = it.attrib['id']
ret.compartment[ it.attrib['id'] ] = it.attrib['compartment']
if "initialAmount" in it.attrib:
vol = ret.env[it.attrib["compartment"]]
key,val = it.attrib['id'] , float(it.attrib['initialAmount'])/vol
ret.env[key] = val
elif "initialConcentration" in it.attrib:
vol = ret.env[it.attrib["compartment"]]
key,val = it.attrib['id'] , float(it.attrib['initialConcentration'])
ret.env[key] = val
else:
pass
for it in root.findall('.//sbml:listOfParameters' , namespaces=ns):
for r in it.getchildren():
if 'value' in r.attrib:
ret.env[ r.attrib['id'] ] = eval(r.attrib['value'])
for it in root.findall('.//sbml:functionDefinition' , namespaces=ns):
fndef = it.find('.//mml:math' , namespaces=ns)
key = it.attrib['id']
if fndef!=None:
fn = parse_math(fndef)
ret.env[key] = fn
listOfInitialAssignments = root.find('.//sbml:listOfInitialAssignments' , namespaces=ns)
if listOfInitialAssignments!=None:
for it in listOfInitialAssignments.getchildren():
varname = it.attrib['symbol']
ret.env[varname] = eval_math(parse_math(it.getchildren()[0]) , ret.env)
listOfRules = root.find('.//sbml:listOfRules' , namespaces=ns)
if listOfRules!=None:
for it in listOfRules.getchildren():
_ , tagname = it.tag.split('}')
key = it.attrib['variable']
if tagname=="assignmentRule":
rule = parse_math(it.getchildren()[0])
ret.rules[key] = rule
else:
ret.args.append( key )
ret.reactions.append( ([] , [key] , parse_math(it.getchildren()[0]) ,{}) )
for r in root.find('.//sbml:listOfReactions' , namespaces=ns).getchildren():
reactants = []
products = []
local_env = {}
listOfReactants = r.find('.//sbml:listOfReactants' , namespaces=ns)
if listOfReactants!=None:
for it in listOfReactants.getchildren():
reactants.append( it.attrib['species'] )
if 'id' in it.attrib:
local_env[ it.attrib['id'] ] = it.attrib['species']
listOfProducts = r.find('.//sbml:listOfProducts' , namespaces=ns)
if listOfProducts!=None:
for it in listOfProducts.getchildren():
products.append( it.attrib['species'] )
if 'id' in it.attrib:
local_env[ it.attrib['id'] ] = it.attrib['species']
listOfModifiers = r.find('.//sbml:listOfModifiers' , namespaces=ns)
if listOfModifiers!=None:
for it in listOfModifiers.getchildren():
if 'id' in it.attrib:
local_env[ it.attrib['id'] ] = it.attrib['species']
listOfLocalParameters = r.find('.//sbml:listOfLocalParameters' , namespaces=ns)
if listOfLocalParameters!=None:
for it in listOfLocalParameters.getchildren():
local_env[ it.attrib['id'] ] = eval(it.attrib['value'])
sm = r.find('.//mml:math' , namespaces=ns)
ret.reactions.append( (reactants , products , parse_math(sm) , local_env) )
return ret
try: #-- python2
from urllib2 import urlopen
except: #-- python3
from urllib.request import urlopen
import matplotlib.pyplot as plt
def runsbml(path , t0 , t1, dt):
try:
root = et.parse(path).getroot()
except:
req = urlopen(path)
s = req.read()
root = et.fromstring(s)
m = readsbml(root)
r = ode(m, None).set_integrator('vode', method='bdf', nsteps=1500 , with_jacobian=False)
r.set_initial_value(m.values() , t0)
res = []
times = []
while r.successful() and r.t < t1:
_ = r.integrate(r.t+dt)
times.append( r.t )
res.append( r.y )
for n,name in enumerate(m.args):
dat = [x[n] for x in res]
plt.plot(times , dat , label=m.namemap[name])
plt.legend()
plt.show()
if __name__=="__main__":
#-- calcium oscillation
runsbml("http://www.ebi.ac.uk/compneur-srv/biomodels-main/download?mid=BIOMD0000000184" , 0.0 , 2000.0 , 0.5)